摘要:研究團(tuán)隊開發(fā)了FINCHES計算框架,利用粗粒化力場預(yù)測內(nèi)在無序區(qū)域與折疊蛋白/其他IDRs的化學(xué)特異性相互作用。
在蛋白質(zhì)王國里,存在著一群特立獨行的"變形者"——內(nèi)在無序區(qū)域(intrinsically disordered regions, IDRs)。這些占人類蛋白質(zhì)組70%以上的區(qū)域雖缺乏固定三維結(jié)構(gòu),卻通過化學(xué)特異性相互作用(chemically specific interactions)調(diào)控著細(xì)胞生命活動。傳統(tǒng)方法難以捕捉這種動態(tài)多變的分子識別過程,而FINCHES計算框架的誕生改變了這一局面。
 圖1 基于序列的由無序區(qū)域驅(qū)動的分子間相互作用預(yù)測
圖1 基于序列的由無序區(qū)域驅(qū)動的分子間相互作用預(yù)測研究團(tuán)隊巧妙改造了粗粒化力場(coarse-grained force fields)的化學(xué)物理參數(shù),將其轉(zhuǎn)化為預(yù)測IDR行為的強(qiáng)大工具。就像破譯分子密碼本,F(xiàn)INCHES能直接從氨基酸序列中解讀出:哪些區(qū)域會與折疊蛋白表面"親密接觸",哪些片段會驅(qū)動液-液相分離(liquid-liquid phase separation),以及磷酸化修飾如何像分子開關(guān)般調(diào)控這些相互作用。
驗證實驗展示了三重預(yù)測威力:準(zhǔn)確識別IDR-IDR互作熱點、量化序列對相分離的調(diào)控、揭示無序區(qū)域與結(jié)構(gòu)化蛋白表面的結(jié)合模式。更令人振奮的是,該方法能在數(shù)分鐘內(nèi)完成全蛋白組掃描,不僅發(fā)現(xiàn)IDR內(nèi)部存在化學(xué)特性迥異的"功能子域",還系統(tǒng)性解析了磷酸化這一關(guān)鍵翻譯后修飾如何重塑相互作用網(wǎng)絡(luò)。
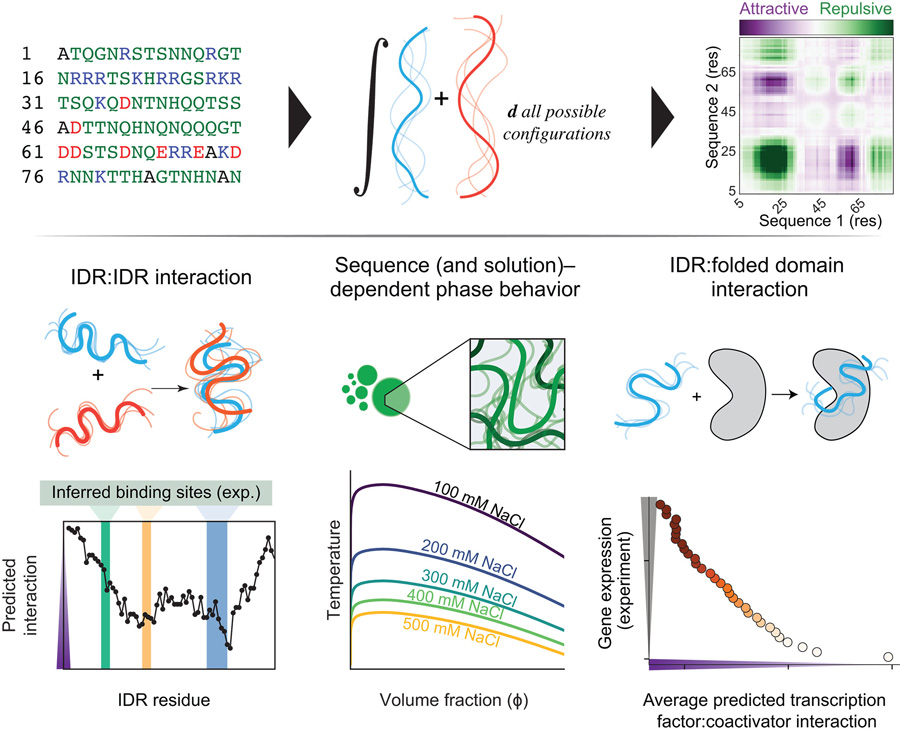
圖2 內(nèi)在無序區(qū)域可通過互補(bǔ)化學(xué)作用驅(qū)動與配體的結(jié)合
盡管無法預(yù)測依賴精確序列的有序界面(如部分折疊后發(fā)生的structured interface),但FINCHES為理解IDR的"模糊邏輯"(fuzzy logic)提供了全新視角。開源工具包和在線服務(wù)器(https://www.finches-online.com/)的開放,將推動更多研究者探索這片充滿可能性的"無序疆域"。
參考資料
[1] Sequence-based prediction of intermolecular interactions driven by disordered regions
摘要:研究團(tuán)隊開發(fā)了FINCHES計算框架,利用粗粒化力場預(yù)測內(nèi)在無序區(qū)域與折疊蛋白/其他IDRs的化學(xué)特異性相互作用。
在蛋白質(zhì)王國里,存在著一群特立獨行的"變形者"——內(nèi)在無序區(qū)域(intrinsically disordered regions, IDRs)。這些占人類蛋白質(zhì)組70%以上的區(qū)域雖缺乏固定三維結(jié)構(gòu),卻通過化學(xué)特異性相互作用(chemically specific interactions)調(diào)控著細(xì)胞生命活動。傳統(tǒng)方法難以捕捉這種動態(tài)多變的分子識別過程,而FINCHES計算框架的誕生改變了這一局面。
 圖1 基于序列的由無序區(qū)域驅(qū)動的分子間相互作用預(yù)測
圖1 基于序列的由無序區(qū)域驅(qū)動的分子間相互作用預(yù)測研究團(tuán)隊巧妙改造了粗粒化力場(coarse-grained force fields)的化學(xué)物理參數(shù),將其轉(zhuǎn)化為預(yù)測IDR行為的強(qiáng)大工具。就像破譯分子密碼本,F(xiàn)INCHES能直接從氨基酸序列中解讀出:哪些區(qū)域會與折疊蛋白表面"親密接觸",哪些片段會驅(qū)動液-液相分離(liquid-liquid phase separation),以及磷酸化修飾如何像分子開關(guān)般調(diào)控這些相互作用。
驗證實驗展示了三重預(yù)測威力:準(zhǔn)確識別IDR-IDR互作熱點、量化序列對相分離的調(diào)控、揭示無序區(qū)域與結(jié)構(gòu)化蛋白表面的結(jié)合模式。更令人振奮的是,該方法能在數(shù)分鐘內(nèi)完成全蛋白組掃描,不僅發(fā)現(xiàn)IDR內(nèi)部存在化學(xué)特性迥異的"功能子域",還系統(tǒng)性解析了磷酸化這一關(guān)鍵翻譯后修飾如何重塑相互作用網(wǎng)絡(luò)。

圖2 內(nèi)在無序區(qū)域可通過互補(bǔ)化學(xué)作用驅(qū)動與配體的結(jié)合
盡管無法預(yù)測依賴精確序列的有序界面(如部分折疊后發(fā)生的structured interface),但FINCHES為理解IDR的"模糊邏輯"(fuzzy logic)提供了全新視角。開源工具包和在線服務(wù)器(https://www.finches-online.com/)的開放,將推動更多研究者探索這片充滿可能性的"無序疆域"。
參考資料
[1] Sequence-based prediction of intermolecular interactions driven by disordered regions



